Neue Einblicke in die DNA-Verpackung
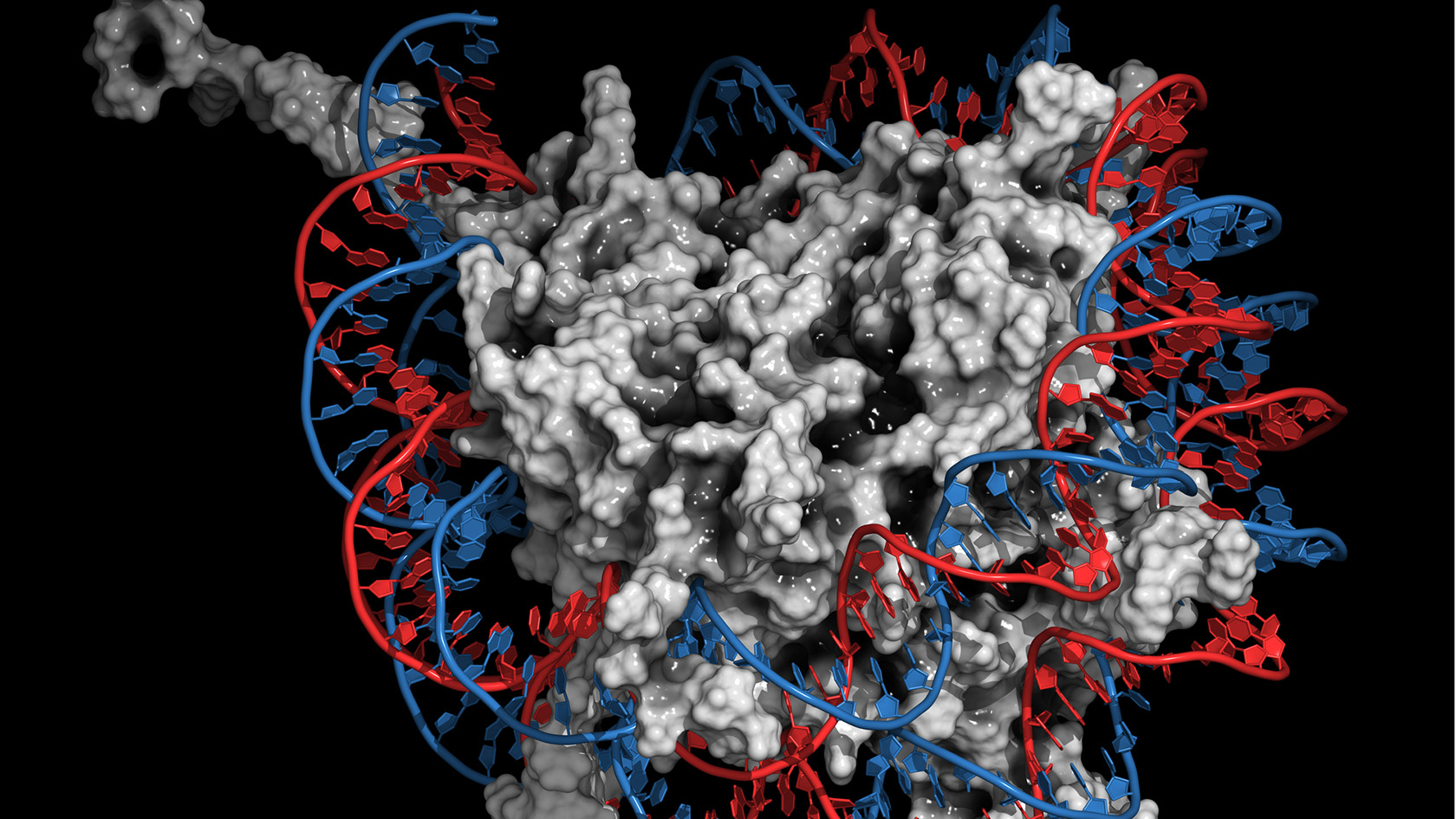
Proteine sind der zentrale Baustoff unseres Körpers und elementarer Bestandteil der Zelle sowie der DNA mit ihren Genen – also dessen, was uns ausmacht. Wissenschaftlerinnen und Wissenschaftler, die die komplexen Wechselwirkungen von Proteinen erforschen, nutzen vermehrt Hochleistungsrechnen (HPC), um im Detail sichtbar zu machen, was im Experiment geschieht. Kürzlich ist es einem internationalen Forscherteam unter der Leitung der Forschungsgruppe von Ben Schuler, Professor für Molekulare Biophysik an der Universität Zürich, gelungen, mit Hilfe von HPC erstmals auf molekularer Ebene den Prozess aufzuschlüsseln, der die DNA im Zellkern aktiviert.
Essenziell ist bei diesem Prozess, dass die Packung der DNA im sogenannten Nukleosom gelockert wird. Das Nukleosom ist eine Einheit aus DNA-Ketten, die um Histone – Proteine, die für die Verpackung wichtig sind – gewickelt sind. Nukleosomen sind die zentralen Bestandteile des Chromatins, in dem die etwa zwei Meter langen Ketten der DNA im Zellkern auf einem winzigem Raum von etwa 1000 Kubikmikrometer komprimiert sind. Dabei hilft ein bestimmtes, stark positiv geladenes Histon (H1), die stark negativ geladene DNA quasi zu versiegeln und zu kompaktieren. Um diese enge Verpackung zu lockern, um dann beispielsweise die DNA zu vervielfältigen oder zu reparieren, muss dieses Histon H1 abgelöst werden.
Hauptakteure: Geladene Proteine
Allerdings: Wie sich die starke Bindung zwischen dem Histon H1 und der DNA in nützlicher Frist aufheben lässt, war bis anhin unklar. Den Forschenden ist es nun gelungen, mit Hilfe von Experimenten in Kombination mit Simulationen auf dem CSCS-Supercomputer «Piz Daint» den Vorgang zu entschlüsseln. Eine zentrale Rolle in dem Prozess nehmen demnach die unstrukturierten Bereiche des Histons H1 sowie eines Proteins namens Prothymosin alpha ein. Dieses Molekül ist – im Gegensatz zum positiv geladenen H1 – stark negativ geladen.
Aufgrund der im Labor durchgeführten Experimente vermuteten die Forschenden bereits, dass neben dem H1 auch Prothymosin alpha entscheidend ist: Die Experimente zeigen, dass Prothymosin alpha mit dem Histon über seine Ladung interagiert und den Ablösungsprozess beschleunigt. Doch erst die Simulationen machten die Details sichtbar. Die Ergebnisse ihre Studie publizierten die Forscher kürzlich in der Fachzeitschrift Nature Chemistry.
Einzigartige Beobachtungen
Die Forschenden visualisierten anhand der Ergebnisse der Simulationen, wie die Proteine Prothymosin alpha und H1 über ihre unstrukturierten Bereiche aneinander binden. Die Visualisierung zeigt, wie Prothymosin alpha, H1 und die DNA kurzfristig einen Komplex bilden. «Das hat in dieser Form noch niemand beobachtet», sagt Schuler. «Diese unstrukturierten Proteine scheinen eine Wechselwirkung zu ermöglichen, die über die traditionelle Strukturbiologie sehr schwierig zu erklären ist.» Konkret schwächt die Verbindung zwischen Prothymosin alpha und H1 die Wechselwirkung zwischen positiv geladenem H1 und negativ geladener DNA, so dass Prothymosin alpha sozusagen die Oberhand gewinnt und das Histon von der DNA ablöst und wegtransportiert: Die DNA ist bereit für die Vervielfältigung oder Reparatur.
Entscheidend für diese Entdeckung war die sogenannte Einzelmolekülspektroskopie, auf die sich die Forschungsgruppe von Schuler spezialisiert hat, in Kombination mit Simulationen. Bis vor etwa zehn Jahren lag der Fokus der Forschung auf der wohldefinierten, gefalteten dreidimensionalen Struktur der Proteine. «Dagegen fanden die unstrukturierten Segmente, die etwa in zwei Dritteln der Proteine unseres Körpers vorkommen, in der traditionellen Strukturbiologie kaum Beachtung», sagt Schuler.
Erst die Spitze des Eisbergs
In der Erforschung der Rolle der unstrukturierten Proteine in zellulären Prozessen seien die nun vorliegenden Erkenntnisse erst die Spitze des Eisbergs, ist Schuler überzeugt. Laut dem Biophysiker zeichnet sich bereits ab, dass es weitere Beispiele gibt, bei denen unstrukturierte geladene Proteine in der Zelle weitere Austauschreaktionen ermöglichen. Schuler und sein Team sind bereits daran, ähnliche Mechanismen, die bei der Reparatur der DNA eine Rolle spielen, zu erforschen.