Genforschung zum Anfassen
Wie isoliert man DNA? Wie zerschneidet man einen Plasmidring? Wie macht man die Erbsubstanz sichtbar? Das «Begreifen, wie Gentechnik funktioniert» – so auch das Motto des vom Verein «Forschung für Leben» organisierten Kurses – löste die drei Fragen in drei Prozessen. Schweizweit finden zurzeit Veranstaltungen statt, um Grundlagen der Genforschung in Dialog und Praxis der Bevölkerung näher zu bringen – ein erfolgreiches Mittel gegen auf Unklarheit basierende Ängste und Skepsis.

Wer es bis zehn Uhr am Vormittag durch das Hörsaallabyrinth der Universität Zürich Irchel mit den auf den ersten Blick kryptischen Raumbezeichnungen zum Raum Y3-L-1834 L02 schaffte, bekam prompt eine Pipette überreicht und wurde kopfüber ins 37° warme Wasser – so die Idealtemperatur für in der Genforschung elementare, chemische Abläufe – geworfen. Doch dank der Anleitung der Biologen Luzia Reutimann und Dominique Grüter ging die bunt gemischte Teilnehmerschar vom Alt-Pathologen bis zur Maturandin nicht zwischen Adenin, Cytosin, Guanin und Thymin unter. «Mehr Wissen über Genetik erfahren», «Endlich das praktisch machen, was man sonst seinen Studenten erklärt» oder «für die Maturaarbeit recherchieren» – nur drei von vielen Gründen, welche rund 20 Neugierige ins Labor lockten.
Auf das Bakterium gekommen
Keine Pflanze «die statt Blütenblätter Stängelblätter hat», wie der Biologe Grüter die Möglichkeiten der Gentechnik erörterte galt es zu schaffen, sondern erstes Ziel war es die DNA «sicherer Escherichia coli» – kurz E. Coli – Bakterien zu isolieren. «An der Luft überleben diese normalerweise im menschlichen Darm lebenden Bakterien nicht lange» beruhigte er weiter. Dennoch, ganz ungefährlich sind sie nicht, da sie starke Toxine enthalten, welche zu Durchfallerkrankungen führen könnten: «Wer etwas verschüttet möge sich melden» und «Handschuhe tragen ist ratsam.»
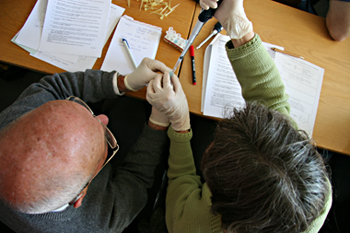
Mit Latex gerüstet und Pipette gewappnet ging es nach kurzer Einführung in medias res. Mit auf den Mikroliter – ein millionstel Liter – genauem Präzisionswerkzeug galt es erst den Nährlösungsübersatz der E.Coli Bakterien abzusaugen. Hierzu war eine ruhige Hand gefragt. Chemische Puffer zerlegten anschliessend die Einzeller. Alles «Uninteressante» – wie Zellwand, Phosphorlipidmembran mit DNA, Ribosome und Ähnliches – wurde durch mehrmaliges Zentrifugieren und (Ab-)pipettieren entfernt. Denn das Interessante sind lediglich die Plasmide – kleine ringförmige DNA-Teile in Bakterien. Nach gut einer Stunde war dann das isolierte Objekt der Begierde im Eppendorf-Röhrchen und die Pause verdient.
Leicht «verdaulich»
Wenn der Molekularbiologe vom «Verdauen» spricht geht es ihm weniger um das gerade eingenommene Mensa- Mittagessen, als um das gezielte Zerschneiden von DNA – der zweite Schritt des Experiments. Dank der aus Bakterien gewonnen Restriktionsenzyme EcoR 1, Bgl 1 und Dde 1 ist es möglich die Kilobasenketten – Adenin-Thymin-Cytosin- Guanin-Ketten, kurz ATGC – der Plasmide punktgenau in Fragmente zu trennen.
Jedes Restriktionsenzym schneidet an unterschiedlichen Stellen und unterschiedlich oft. EcoR1 einmal – mit dem Ergebnis, dass aus dem Plasmidring eine lange Kette wird. Bgl 1 schneidet zweimal, folglich bleiben zwei kürzere Kettenteile und Dde 1 schneidet sechsmal, was sechs winzig kleine Fragmente ergibt. Den sichtbaren Beweis für die «Schnitte» sollten wir erst später bekommen. Wer einen fast unsichtbaren Mikroliter Dde 1 pipettiert und verständlicherweise euphorisch «Voller Erfolg» ruft fragt sich sicher: Wozu der ganze Aufwand?

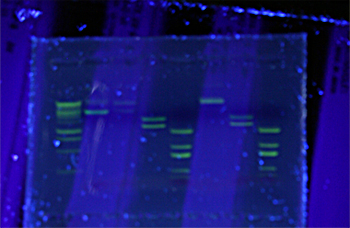
Darum: Im letzten Prozess – der Gel-Elektrophorese – lässt man die negativ geladenen Plasmid-DNA-Fragmente mit Hilfe von rund 200 Volt in einem Gel-Behälter vom Minuspol zum Pluspol wandern. Logischerweise sind kleine Fragmente schneller als grosse und so wurde nach einer halben Stunde mit Hilfe eines teuren Farbstoffes namens SYBR green und UV-Licht das sichtbar, was wir eigentlich erwarteten: die DNA-Kilobasenketten.
Genforschung für das Volk
Bei der zunehmenden Entfremdung und Entfernung der Forschung von der Bevölkerung ist dieses «Genlabor für Erwachsene» ein Beispiel das Schule machen könnte – und auch macht. «Regelmässig bieten wir Kurse für Schüler im Maturajahr an», so Reutimann, und man stösst auch hier auf breites Interesse – kein Wunder, denn: «Oft haben weder die Lehrer das Know-how, noch die Schulen die Ausrüstung derartige Experimente durchzuführen».
Zahlreiche Fragen, auch ethischer Natur waren Kern der abschliessenden Diskussion – und es wurde klar, dass weitere derartige Veranstaltungen nötig wären, um noch mehr Klarheit zu schaffen. Es gibt aber nur wenige Forschende, die sich die Mühe der Vermittlung machen. Während die fachliche «Herausforderung der Molekularbiologie» laut Grüter darin liegt, herauszufinden «welche exakte Funktion welches Gen hat», so besteht ihr gesellschaftlicher Auftrag darin, bei all der Angst gegen «Genmais», «Designerkind» und dem «Klonen» zumindest die Grundlagen ihres Fachbereichs anschaulich zu machen.